根据他们的Github页面,Neo4j Runway 是一个 Python 库,简化了将关系型数据迁移到图数据库的过程。它提供了抽象与 OpenAI 通信的工具,以发现数据并生成数据模型,还提供了生成导入代码并加载数据到 Neo4j 实例的工具。通过上传一个 CSV 文件,大型语言模型会自动识别节点和关系,并生成知识图谱。最近我看到一篇帖子提到了neo4j-runway。
知识图在医疗领域中是一种强大的工具,用于组织和分析复杂的医疗数据。这些图以更直观的方式展示不同实体之间的关系,如疾病、治疗、患者和医疗服务提供者。
KG在医疗领域有一些有用的应用:
-
多种数据源的集成: 知识图谱可以整合来自各种来源的数据,包括电子健康记录(EHR)、医学研究论文、临床试验结果、基因组数据和患者病史。
-
改善临床决策支持: 通过将症状、诊断、治疗和结果联系起来,知识图谱可以增强临床决策支持系统(CDSS),鉴于它们考虑了大量的相互关联的医学知识,这可能提高诊断准确性并增强治疗效果。本文将探讨这一话题。
-
个性化医疗: 知识图谱能够帮助开发个性化的治疗计划,通过将患者特定的数据与更广泛的医学知识相关联。这包括理解遗传信息、疾病机制与治疗反应之间的关系,从而实现更个性化的医疗干预。
-
药物发现与开发: 在药物研究中,知识图谱可以通过识别潜在的药物靶点及了解疾病涉及的生物学途径来加速药物发现。
- 公共卫生与流行病学: 知识图谱在公共卫生中对于追踪疾病爆发、理解流行病学趋势以及规划干预措施很有用,因为它们可以整合来自各种公共卫生数据库、社交媒体和其他来源的数据,从而提供对公共健康威胁的实时洞察。
Neo4j Runway 是由 Alex Gilmore 开发的一个开源库。你可以在 这里 找到它的仓库,并在 这篇博客 中了解更多关于它的信息。
目前,此库目前仅支持OpenAI的大型语言模型来解析CSV文件,提供以下功能:
- 数据发现:利用OpenAI大模型从您的数据中提取有价值的见解。
- 图数据建模:使用OpenAI和Instructor Python库创建准确的图数据模型。
- 代码生成:生成适合您数据加载需求的代码。
- 数据导入:使用Runway内置的PyIngest实现(一种广泛使用的Neo4j数据导入工具)加载您的数据。
- 无需担心Cypher编写, 因为大语言模型会处理所有这些任务。
在这里,除了让LLM完成所有CSV到知识图谱的转换,我还使用了Langchain的_GraphCypherQAChain_作为最后一步,从提示中生成Cypher查询语句,这样我们就可以在不编写任何Cypher(用于查询Neo4j图数据库的语言)的情况下查询图。
该库的GitHub页面有一个金融例子,但我更想看看它在医疗场景中是否好用。从Kaggle上的一个非常简单的数据集入手(疾病症状和患者资料数据集),这个数据集只包含10个字段(疾病、发热、咳嗽、疲劳、呼吸不畅、年龄、性别、血压、胆固醇和结果变量),我希望能够把这些数据提供给大模型以获得诊断建议。
那么,我们直接来看代码吧,首先,加载库:
sudo apt install python3-pydot graphviz安装Python3的pydot和graphviz库。
命令如下:
pip install neo4j-runway# 引入numpy库
import numpy as np
# 引入pandas库
import pandas as pd
# 引入neo4j_runway模块中的Discovery、GraphDataModeler、IngestionGenerator、LLM和PyIngest类
from neo4j_runway import Discovery, GraphDataModeler, IngestionGenerator, LLM, PyIngest
# 引入IPython.display模块中的display、Markdown和Image类
from IPython.display import display, Markdown, Image加载环境变量部分:你可以参考我另一篇文章以了解如何在Neo4j Aura中创建一个实例并验证身份。
首先,加载环境变量:load_dotenv()。然后,从环境变量中获取OPENAI_API_KEY。接着,获取NEO4J_URL。最后,从环境变量中获取NEO4J_PASSWORD。这是加载环境变量并从环境变量中获取API密钥和数据库连接信息的代码示例。
load_dotenv()
OPENAI_API_KEY = os.getenv('sk-openaiapikeyhere')
NEO4J_URL = os.getenv('neo4j+s://your.databases.neo4j.io')
NEO4J_PASSWORD = os.getenv('yourneo4jpassword')
我们现在来加载医学数据。从[Kaggle网站](https://www.kaggle.com/datasets/uom190346a/disease-symptoms-and-patient-profile-dataset)下载CSV数据文件,并在Jupyter笔记本中加载。这其实是一个相当简单的数据集,但很有用,可以用来测试一个概念,非常实用。
disease_df = pd.read_csv('/home/user/Disease_symptom.csv')
# 这行代码读取了一个CSV文件,包含了疾病和症状的数据,并将其存储为一个名为'disease_df'的DataFrame。
# Zhè háng dǎnmǎ dúqǔ le yīgè CSV wénjiàn, bāochánɡ le jíbìng hé zhēngzhàng de shùjù, bìng jiāqí cúnchǔ wéi yīgè mìngmínɡ 'disease_df' de DataFrame.
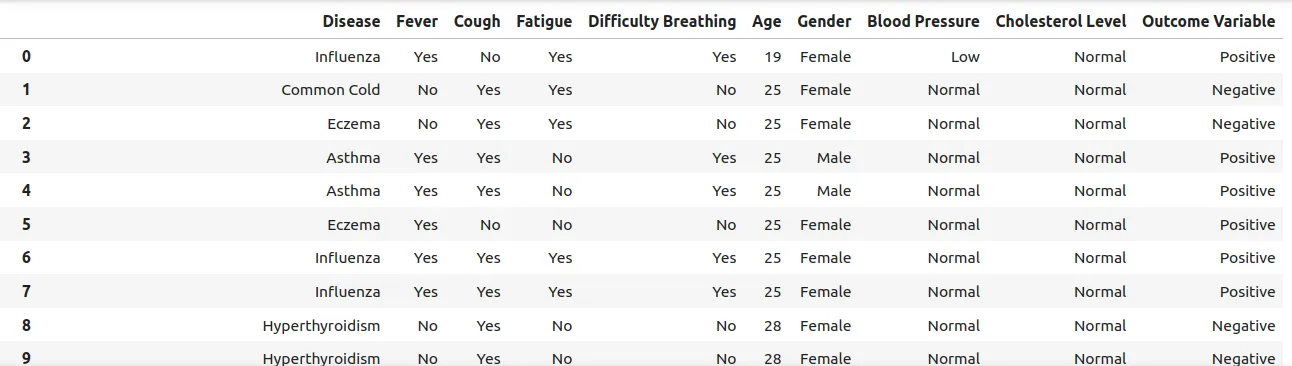
例如,我们可以列出所有引起呼吸困难的疾病,这不仅有助于在图中选择节点,还有助于形成诊断假设。
disease_df[disease_df['困难呼吸']=='是']
让我们继续。所有变量都必须是字符串(这个库就是这么设计的),即使是整数型也一样。接着,我们就保存 CSV 文件:
# 移除疾病数据框列名中的多余空格
disease_df.columns = disease_df.columns.str.strip()
# 遍历疾病数据框的每一列
for i in disease_df.columns:
# 将疾病数据框中的每一列转换为字符串类型
disease_df[i] = disease_df[i].astype(str)
# 将处理后的疾病数据框保存为CSV文件,不包含索引
disease_df.to_csv('/home/user/disease_prepared.csv', index=False)
接下来,我们将描述将提供给LLM的数据,包括每个字段可能的值:
DATA_DESCRIPTION = {
'Disease': '疾病的名称或医疗状况。',
'Fever': '患者是否发热(是/否)。',
'Cough': '患者是否有咳嗽症状(有/无)。',
'Fatigue': '患者是否感到疲劳(是/否)。',
'Difficulty Breathing': '患者是否有呼吸困难(是/否)。',
'Age': '患者的年龄(岁)。',
'Gender': '患者的性别(男/女)。',
'Blood Pressure': '患者的血压水平(正常/高)。',
'Cholesterol Level': '患者的胆固醇水平(正常/高)。',
'Outcome Variable': '表示特定疾病诊断或评估结果的结局变量(阳性/阴性)。'
}
下一步是让LLM分析表格中的数据,找出对生成图表数据模型重要的数据元素。
disc = Discovery(llm=llm, user_input=DATA_DESCRIPTION, data=disease_df) # Discovery类的实例化,其中llm、DATA_DESCRIPTION和disease_df是输入参数
disc.run() # 调用实例的run方法
这会生成数据分析的Markdown格式输出。

好的,现在我们来创建初始模型。
# 创建图数据建模器实例
gdm = GraphDataModeler(llm=llm, discovery=disc)
# 生成模型
gdm.create_initial_model()
# 可视化数据模型
gdm.current_model.visualize()

在这里,重点在疾病上,所以咱们就来调整一下某些关系。
gdm.iterate_model(user_corrections='''
让我们一步一步来思考。请对数据模型进行以下更新: -
删除Patient与Disease之间的关系,删除Patient与Symptom之间的关系,以及删除Patient与Outcome之间的关系。
-
将Patient节点更改为Demographics。
-
从Disease节点到Demographics节点创建HAS_DEMOGRAPHICS关系。
-
从Disease节点到Symptom节点创建HAS_SYMPTOM关系。如果Symptom值为'无',则删除此关系。
-
从Disease节点到HealthIndicator节点创建HAS_LAB关系。
-
从Disease节点到Outcome节点创建HAS_OUTCOME关系。
''')from IPython.display import Image, display
显示一个特定宽度的图像
gdm.current_model.visualize().render('output', format='png')img = Image('output.png', width=1200) # 根据需要调整
display(img)
现在我们可以生成Cypher代码和YAML文件,然后将数据加载到Neo4j中。如果你只是在测试或第二次这样做,你可能想将实例清空,以清除所有数据。
# 实例化导入生成器
gen = IngestionGenerator(data_model=gdm.current_model,
username="neo4j",
password='yourneo4jpasswordhere',
uri='neo4j+s://123654888.databases.neo4j.io',
database="neo4j",
csv_dir="/home/user/",
csv_name="disease_prepared.csv")
# 生成导入YAML文件
pyingest_yaml = gen.generate_pyingest_yaml_string()
# 保存YAML文件
gen.generate_pyingest_yaml_file(file_name="disease_prepared")一切都准备好了,咱们来把数据导入实例吧。
PyIngest(yaml_string=pyingest_yaml, dataframe=disease_df)访问 Neo4j Aura 实例 ,Open,输入你的密码,并在此处运行该 Cypher 查询。
匹配 (n)
WHERE n:人口统计 OR n:疾病 OR n:症状 OR n:结果 OR n:健康指标
可选匹配 (n)-[r]->(m)
返回 n, r, m按 Ctrl + Enter,你就可以看到这样的结果:
检查节点和它们之间的关系后,我们发现症状、健康指标和人口统计信息之间存在着巨大的相互关联。
让我们来看看糖尿病:由于没有应用任何筛选条件,会显示男性和女性,以及所有LAB、DEMOGRAPHIC和OUTCOME的选项(注:LAB指实验室检查,DEMOGRAPHIC指人口统计学,OUTCOME指结果)。
MATCH (n:疾病 {name: '糖尿病'})
WHERE n 是人口统计 OR n 是疾病 OR n 是症状 OR n 是结果 OR n 是健康指标
OPTIONAL MATCH (n)-[r]->(m)
RETURN n, r, m或者所有这些在临床检查时出现高血压的病症:
// 匹配疾病节点
MATCH (d:Disease)
// 匹配疾病节点到实验室节点的HAS_LAB关系
MATCH (d)-[r:HAS_LAB]->(l)
// 匹配疾病节点到结果节点的HAS_OUTCOME关系
MATCH (d)-[r2:HAS_OUTCOME]->(o)
// 确保实验室节点的血压属性设置为'High'
WHERE l.血压 = 'High' AND o.result='阳性'
RETURN d, properties(d) AS disease_properties, r, properties(r) AS relationship_properties, l, properties(l) AS lab_properties现在我的目标已经很清楚,即:我想将一份医疗报告提交给一个LLM,即Google的 Gemini-1.5-Flash,通过Langchain(GraphCypherQAChain)自动生成Cypher查询语句,根据患者的症状、健康指标等信息,返回可能的疾病。那就开始吧:
import warnings
import json
from langchain_community.graphs import Neo4jGraph
# 忽略所有警告
with warnings.catch_warnings():
warnings.simplefilter('ignore')
# Neo4j 用户名
NEO4J_USERNAME = "neo4j"
# Neo4j 数据库名称
NEO4J_DATABASE = 'neo4j'
# Neo4j 连接 URI
NEO4J_URI = 'neo4j+s://1236547.databases.neo4j.io'
# Neo4j 密码
NEO4J_PASSWORD = 'yourneo4jdatabasepasswordhere'
# 注意:请勿在此处硬编码实际密码从实例对象和模式定义中获取知识图谱:这里包括节点属性和关系属性。
kg = Neo4jGraph(
url=NEO4J_URI, username=NEO4J_USERNAME, password=NEO4J_PASSWORD, database=NEO4J_DATABASE
)
kg.refresh_schema()
print(textwrap.fill(kg.schema, 60))
schema = kg.schema 咱们来启动Vertex AI Gemini-1.5-Flash:
from langchain.prompts.prompt import PromptTemplate
from langchain.chains import GraphCypherQAChain
from langchain.llms import VertexAI
# 初始化Vertex AI服务
vertexai.init(project="your-project", location="us-west4")
llm = VertexAI(model='gemini-1.5-flash')现在,最难的部分来了:为Gemini-1.5-Flash制定详细的指示,使其能够自动生成查询图数据库的密语查询,以便我们得到所需的结果。我们需要一个MASTER提示来完成这个任务! 😂😂😂😂(逻辑推理+少量示例提示)
prompt_template = """
让我们一步一步思考:
步骤1:任务:
生成一个有效且简洁的Cypher语句来查询图数据库,语句长度应少于256个字符。
步骤2:了解数据库模式: {schema}
步骤3:指令:
- 在Cypher查询中,仅使用提供的关系类型和属性,这些关系类型和属性必须同时出现在模式和用户问题中。
- 在Cypher查询中,不使用用户问题中提供的任何关系类型或属性,如果这些关系类型或属性未在提供的模式中包含。
- 对于年龄,绝不要直接使用具体年龄值。例如,不要使用‘24岁’,而应当使用区间表示法,如‘大于20岁’。
- 只使用‘大于’条件来表示年龄,不要使用‘小于’或‘等于’。
- 不要使用数据库中不存在的属性键。
步骤4:示例:
以下是一些针对特定问题生成的Cypher语句示例:
4.1 哪些疾病表现出高血压?
MATCH (d:Disease)
MATCH (d)-[r:HAS_LAB]->(l)
WHERE l.bloodPressure = 'High'
RETURN d.name
4.2 哪些疾病表现出高血压的症状?
MATCH (d:Disease)
MATCH (d)-[r:HAS_LAB]->(l)
MATCH (d)-[r2:HAS_OUTCOME]->(o)
WHERE l.bloodPressure = 'High' AND o.result='Positive'
RETURN d, properties(d) AS disease_properties, r, properties(r) AS relationship_properties, l, properties(l) AS lab_properties
4.3 老年人中,患者表现出高血压、高胆固醇、发热、疲劳的疾病的名称是什么?
MATCH (d:Disease)
MATCH (d)-[r1:HAS_LAB]->(lab)
MATCH (d)-[r2:HAS_SYMPTOM]->(symptom)
MATCH (symptom)-[r3:HAS_DEMOGRAPHICS]->(demo)
WHERE lab.bloodPressure = 'High' AND lab.cholesterolLevel = 'High' AND symptom.fever = 'Yes' AND symptom.fatigue = 'Yes' AND TOINTEGER(demo.age) >40
RETURN d.name
4.4 对于高胆固醇的人群,哪些疾病会表现出发热、疲劳、无咳嗽、无呼吸困难的症状?
MATCH (d:Disease)-[r:HAS_SYMPTOM]->(s:Symptom)
WHERE s.fever = 'Yes' AND s.fatigue = 'Yes' AND s.difficultyBreathing = 'No' AND s.cough = 'No'
MATCH (d:Disease)-[r1:HAS_LAB]->(lab:HealthIndicator)
MATCH (d)-[r2:HAS_OUTCOME]->(o:Outcome)
WHERE lab.elysterolLevel='High' AND o.result='Positive'
RETURN d, properties(d) AS disease_properties, r, properties(r) AS relationship_properties
步骤5. 各实体允许的值如下:
- 发烧:表示患者是否发烧(是/否)。
- 咳嗽:表示患者是否咳嗽(是/否)。
- 疲劳:表示患者是否疲劳(是/否)。
- 呼吸困难:表示患者是否有呼吸困难(是/否)。
- 年龄:患者的具体年龄(以年为单位)。
- 性别:患者的性别(男/女)。
- 血压:患者的血压水平(正常/高血压)。
- 胆固醇水平:患者的胆固醇水平(正常/高)。
- 结果变量:表示特定疾病诊断或评估结果的变量(阳性/阴性)。
步骤6. 回答问题 {question}
""" 我们搭建了GraphCypherQAChain……
cypher_prompt = PromptTemplate(
input_variables=["schema","question"],
template=prompt_template
)
cypherChain = GraphCypherQAChain从_llm(
VertexAI(温度=0.1),
graph=kg,
verbose=True,
cypher_prompt=cypher_prompt,
top_k=10 # 这个值也可以根据需要进行调整
)并提交医疗报告:
cypherChain.run("""
患者信息:
简多伊,一位58岁的女性,于2024年6月15日入院。
主诉与现病史:
简报告称,她在入院前五天开始出现高达104°F的高烧、全身痛和皮疹。
既往史:
简没有显著的既往病史,也没有已知的过敏史。
体格检查:
简的体温为102.8°F,心率为每分钟110次,血压为100/70 mmHg,呼吸频率为每分钟20次/分。未见皮下出血点或紫癜。
她可能患有什么病?""")输出:这里 Gemini-.5-Flash 生成用于查询图形数据库的加密查询,并通过 JSON 将结果返回给 LLM,LLM 解释结果并返回易于理解的响应:
这个结果不包括_Gemini-1.5-Flash_的知识库,而仅仅是在查询的知识图谱。想象一下如果我们有一个包含300个特征的漂亮数据集!
我们可以把 GraphCypherQAChain 中的 _topk 参数调整为 1 或其他任意数值。
如果我们运行最后一个查询,我们将得到77种具有这些症状的疾病的列表,但top_k(指返回的结果数量)被设置为1:
当前的 neo4j-runway 项目处于测试版,并有以下限制:
- 仅支持单一 CSV 输入来生成数据模型
- 节点只能有一个标签
- 仅支持唯一性和节点/关系键约束
- 关系不允许多条唯一性约束
- 生成模型时,不支持引用相同节点属性的 CSV 列
- 目前仅能使用 OpenAI 提供的模型
- Runway 包含的修改版 PyIngest 函数仅能加载本地的 Pandas 数据框或 CSV 文件
感谢
✨ Google 机器学习开发者计划和项目以及 Google Cloud 技术专家计划的创新者项目通过提供 Google Cloud 积分支持了这项工作 ✨
🔗 https://developers.google.com/machine-learning 谷歌机器学习开发者文档