回顾了下PCA的步骤,并用python实现。深刻的发现当年学的特征值、特征向量好强大。
Introduction to PCA
PCA是一种无监督的学习方式,是一种很常用的降维方法。在数据信息损失最小的情况下,将数据的特征数量由n,通过映射到另一个空间的方式,变为k(k<n)。
Sample Data
这里用一个2维的数据来说明PCA,选择2维的数据是因为2维的比较容易画图。
这是数据:

data
import numpy as np x=np.array([2.5,0.5,2.2,1.9,3.1,2.3,2,1,1.5,1.1]) y=np.array([2.4,0.7,2.9,2.2,3,2.7,1.6,1.1,1.6,0.9]) data=np.matrix([[scaled_x[i],scaled_y[i]] for i in range(len(scaled_x))])
Step 1: 求平均值以及做normalization
mean_x=np.mean(x) mean_y=np.mean(y) scaled_x=x-mean_x scaled_y=y-mean_y data=np.matrix([[scaled_x[i],scaled_y[i]] for i in range(len(scaled_x))])
画个图看看分布情况:
import matplotlib.pyplot as plt plt.plot(scaled_x,scaled_y,'o')

data distribution
Step 2: 求协方差矩阵(Covariance Matrix)
协方差矩阵
方差的定义为:

假设n为数据的特征数,那么协方差矩阵M, 为一个nn的矩阵,其中Mij为第i和第j个特征的协方差,对角线是各个特征的方差。
在我们的数据中,n=2,所以协方差矩阵是22的,
通过numpy我们可以很方便的得到:
cov=np.cov(scaled_x,scaled_y)
得到cov的结果为:
array([[ 0.61655556, 0.61544444],
[ 0.61544444, 0.71655556]])
散度矩阵 Scatter Matrix
另一种选择是用scatter matrix,定义为:

sactter matrix
由于我们之前已经做过normalization,因此对于我们来说,
这个矩阵就是 data*data的转置矩阵。
np.dot(np.transpose(data),data)
得到结果:
matrix([[ 5.549, 5.539],
[ 5.539, 6.449]])
我们发现,其实协方差矩阵和散度矩阵关系密切,散度矩阵 就是协方差矩阵乘以(总数据量-1)。因此他们的特征根和特征向量是一样的。这里值得注意的一点就是,散度矩阵是SVD奇异值分解的一步,因此PCA和SVD是有很大联系的,他们的关系这里就不详细谈了,以后有机会再写下。
Step 3: 求协方差矩阵的特征根和特征向量
用numpy计算特征根和特征向量很简单,
eig_val, eig_vec = np.linalg.eig(cov)
但是他们代表的意义非常有意思,让我们将特征向量加到我们原来的图里:
plt.plot(scaled_x,scaled_y,'o',) xmin ,xmax = scaled_x.min(), scaled_x.max() ymin, ymax = scaled_y.min(), scaled_y.max() dx = (xmax - xmin) * 0.2 dy = (ymax - ymin) * 0.2 plt.xlim(xmin - dx, xmax + dx) plt.ylim(ymin - dy, ymax + dy) plt.plot([eig_vec[:,0][0],0],[eig_vec[:,0][1],0],color='red') plt.plot([eig_vec[:,1][0],0],[eig_vec[:,1][1],0],color='red')

data distribution with eig_vec
其中红线就是特征向量。有几点值得注意:
特征向量之间是正交的,PCA其实就是利用特征向量的这个特点,重新构建新的空间体系
特征向量代表着数据的pattern(模式),比如一条代表着y随着x的增大而增大的趋势,而另外一条,则是代表数据也有该方面的变化。所以特征向量的命名是很科学的,他代表着矩阵的特征。
如果我们将数据直接乘以特征向量矩阵,那么其实我们只是以特征向量为基底,重新构建了空间,画个图,感受下吧:new_data=np.transpose(np.dot(eig_vec,np.transpose(data)))
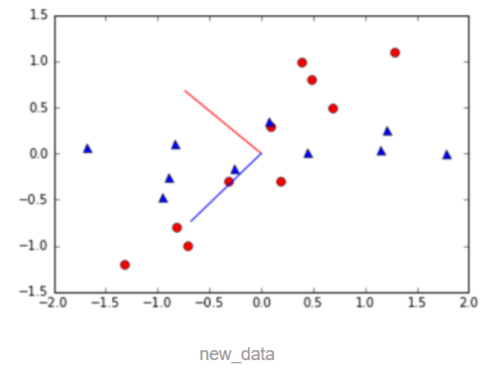
new_data
蓝色的三角形就是经过坐标变换后得到的新点,其实他就是红色原点投影到红线、蓝线形成的。
Step 4: 选择主要成分
得到特征值和特征向量之后,我们可以根据特征值的大小,从大到小的选择K个特征值对应的特征向量。
这个用python的实现也很简单:
eig_pairs = [(np.abs(eig_val[i]), eig_vec[:,i]) for i in range(len(eig_val))] eig_pairs.sort(reverse=True)
从eig_pairs选取前k个特征向量就行。这里,我们只有两个特征向量,选一个最大的。
feature=eig_pairs[0][1]
Step 5: 转化得到降维的数据
主要将原来的数据乘以经过筛选的特征向量组成的特征矩阵之后,就可以得到新的数据了。
new_data_reduced=np.transpose(np.dot(feature,np.transpose(data)))
output:

new_data
数据果然变成了一维的数据。
最后我们通过画图来理解下数据经过PCA到底发生了什么。
plt.plot(scaled_x,scaled_y,'o',color='red') plt.plot([eig_vec[:,0][0],0],[eig_vec[:,0][1],0],color='red') plt.plot([eig_vec[:,1][0],0],[eig_vec[:,1][1],0],color='blue') plt.plot(new_data[:,0],new_data[:,1],'^',color='blue') plt.plot(new_data_reduced[:,0],[1.2]*10,'*',color='green')

final_result
绿色的五角星是PCA处理过后得到的一维数据,为了能跟以前的图对比,将他们的高度定位1.2,其实就是红色圆点投影到蓝色线之后形成的点。这就是PCA,通过选择特征根向量,形成新的坐标系,然后数据投影到这个新的坐标系,在尽可能少的丢失信息的基础上实现降维。
总结
通过上述几步的处理,我们简单的实现了PCA第一个2维数据的处理,但是原理就是这样,我们可以很轻易的就依此实现多维的。
PCA的python3实现
import numpy as npdef pca(X,k):#k is the components you want #mean of each feature n_samples, n_features = X.shape mean=np.array([np.mean(X[:,i]) for i in range(n_features)]) #normalization norm_X=X-mean #scatter matrix scatter_matrix=np.dot(np.transpose(norm_X),norm_X) #Calculate the eigenvectors and eigenvalues eig_val, eig_vec = np.linalg.eig(scatter_matrix) eig_pairs = [(np.abs(eig_val[i]), eig_vec[:,i]) for i in range(n_features)] # sort eig_vec based on eig_val from highest to lowest eig_pairs.sort(reverse=True) # select the top k eig_vec feature=np.array([ele[1] for ele in eig_pairs[:k]]) #get new data data=np.dot(norm_X,np.transpose(feature)) return data
用sklearn的PCA与我们的pca做个比较:
from sklearn.decomposition import PCA X = np.array([[-1, 1], [-2, -1], [-3, -2], [1, 1], [2, 1], [3, 2]]) pca=PCA(n_components=1) pca.fit(X) pca.transform(X)
得到结果:
array([[-0.50917706], [-2.40151069], [-3.7751606 ], [ 1.20075534], [ 2.05572155], [ 3.42937146]])
用我们的pca试试
pca(X,1)
得到结果:
array([[-0.50917706], [-2.40151069], [-3.7751606 ], [ 1.20075534], [ 2.05572155], [ 3.42937146]])
完全一致,完美~
值得一提的是,sklearn中PCA的实现,用了部分SVD的结果,果然他们因缘匪浅。
To Be Continued:
The relationship between PCA and SVD
The implementation of PCA, and the comparison between the results of my PCA and sklearn's PCA (completed)
作者:JxKing
链接:https://www.jianshu.com/p/4528aaa6dc48


 随时随地看视频
随时随地看视频




