无监督领域有一个准度和效率双佳的异常点检测算法,我在实践中使用过几次,效果奇好,就是最近几年非常流行的isolation forest(孤立森林)。该算法在sklearn中有现成的包,但是如果大数据的集群上跑的话,目前没有封装好的接口,给分布式任务的部署带来了很多不便(话说spark mllib中集成的算法真心太少了),本文用scala从头进行该算法在spark上的分布式实现,并演示任务在集群上的执行全过程。
一、算法简介
先说一下算法的最少必要知识,细节部分会揉在代码里进行讲解。
1、训练过程:构建森林的树木
iForest由iTree组成。构建每一颗iTree时,从训练数据中抽取N个样本,然后在这些样本中,随机选择一个特征,再随机选择该特征下的一个值,对样本进行二叉划分,然后分别在左右两边的数据集上重复上面的过程,直接达到终止条件,一颗树构建完成。
2、预测过程:计算样本的异常得分
把测试数据在每棵树上沿对应的条件分支往下走,直到达到叶子节点,并记录这过程中经过的路径长度path length(用h(x)表示)。并由此得出异常分数,当分数超过某一阈值,即可判定为异常样本。
二、scala实现
代码主体非原创,参考自国外的一位大神:https://github.com/hsperr/first_steps_in_scala,有部分修改
1、首先,import编写spark程序所需的包,以及scala的Random模块,用于随机选取功能。
import org.apache.spark.SparkContext._import org.apache.spark.rdd.RDDimport org.apache.spark.{SparkConf, SparkContext}import scala.util.Random2、定义单颗树iTree,第二、三行意味着,每棵树的左右分支ITreeBranch和叶子节点ITreeLeaf都属于iTree的子类。
sealed trait ITreecase class ITreeBranch(left: ITree, right: ITree, split_column: Int, split_value: Double) extends ITreecase class ITreeLeaf(size: Long) extends ITree
3、定义孤立森林的类,完成算法的训练部分,即全部树的构建。
3.1、从样本中抽样,用于构建单个iTree
object IsolationForest { def getRandomSubsample(data: RDD[Array[Double]], sampleRatio: Double, seed: Long = Random.nextLong): RDD[Array[Double]] = {
data.sample(false, sampleRatio, seed=seed)
}3.2 、递归构建生成单颗iTree。
参数:
data:上步抽出的样本数据;
maxHeight:树的最大高度即树终止生长的条件;
numColumns:data的特征数量;
currentHeight:树的当前高度。
返回:
一颗完整的ITree
def growTree(data: RDD[Array[Double]], maxHeight:Int, numColumns:Int, currentHeight:Int = 0): ITree = {
val numSamples = data.count() //递归终止条件,当前树高大于maxHeight或数据量不大于1
if(currentHeight>=maxHeight || numSamples <= 1){ return new ITreeLeaf(numSamples)
} //随机选择特征列
val split_column = Random.nextInt(numColumns)
val column = data.map(s => s(split_column)) //随机选择该特征列中的值split_value,用于分割样本
val col_min = column.min()
val col_max = column.max()
val split_value = col_min + Random.nextDouble()*(col_max-col_min) //小于分割值的成为左子树,反之右子树
val X_left = data.filter(s => s(split_column) < split_value).cache()
val X_right = data.filter(s => s(split_column) >= split_value).cache() //递归
new ITreeBranch(growTree(X_left, maxHeight, numColumns, currentHeight + 1),
growTree(X_right, maxHeight, numColumns, currentHeight + 1),
split_column,
split_value)
}
}3.3、将多棵iTree组建成完整森林iforest
参数:
data:全部训练数据;
numTrees:森林中树的个数;
subSampleSize:每棵树采样的大小;
seed:随机种子。
返回:
孤立森林
def buildForest(data: RDD[Array[Double]], numTrees: Int = 2, subSampleSize: Int = 256, seed: Long = Random.nextLong) : IsolationForest = {
val numSamples = data.count()
val numColumns = data.take(1)(0).size
val maxHeight = math.ceil(math.log(subSampleSize)).toInt
val trees = Array.fill[ITree](numTrees)(ITreeLeaf(1))
val trainedTrees = trees.map(s=>growTree(getRandomSubsample(data, subSampleSize/numSamples.toDouble, seed), maxHeight, numColumns))
IsolationForest(numSamples, trainedTrees)
}4、定义预测功能类
4.1 预测功能类定义为IsolationForest的样例类,
参数
num_samples:单课iTree的样本数目
trees:已经构建好的孤立森林iforest
主函数predict,
参数x:要预测的单条样本数组,
返回:异常得分Anomaly Score
步骤:
在每一棵iTree上,计算样本达到叶子节点走过的路径长度,然后将得到的不同路径长度按照如下公式进行计算,得到异常得分,走过的路径越短,得分越高,代表越异常。
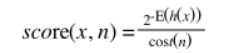
image.png
公式中,h(x)代表路径长度,E(h(x))代表在不同的iTree上路径长度的均值,即群体决策,分母是用来归一化的。
case class IsolationForest(num_samples: Long, trees: Array[ITree]) { def predict(x:Array[Double]): Double = {
val predictions = trees.map(s => pathLength(x, s, 0)).toList println(predictions.mkString(","))
math.pow(2, -(predictions.sum/predictions.size)/cost(num_samples)) //Anomaly Score
}上面代码用到的cost 方法和pathLength方法定义如下,
cost方法参数为二叉树中的样本个数,范围该二叉树的平均路径长度,公式为:
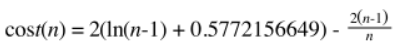
image.png
def cost(num_items:Long): Int = //二叉搜索树的平均路径长度。0.5772156649:欧拉常数 (2*(math.log(num_items-1) + 0.5772156649)-(2*(num_items-1)/num_items)).toInt
pathLength方法是一个递归计算,因为每走一步,接下来面对的仍然是一颗树,分支树或者叶子节点。
参数:样本x,单颗树tree,当前的路径长度path_length,初始值应传入0。
返回:最终的路径长度
@scala.annotation.tailrec final def pathLength(x:Array[Double], tree:ITree, path_length:Int): Double ={
tree match{ //match方法,让tree进行如下两种模式匹配
//如果ITree匹配到的类型是叶子节点,那么,查看该节点的样本数size,如果size大于1,则加上该size对应的二叉搜索树的平均路径长度,如果size等于1,则直接加1
case ITreeLeaf(size) =>
if (size > 1)
path_length + cost(size) else
path_length + 1
//如果ITree匹配到的类型是一颗分支子树,该子树还会有left分支,right分支,以及分类的依据特征列split_column,和该特征列的分割值split_value
case ITreeBranch(left, right, split_column, split_value) =>
val sample_value = x(split_column) //传入的样本x在该特征上的取值
if (sample_value < split_value) //如果小于分割值则在左子树上进行递归计算,如果大于分割值则在右子树上进行递归计算
pathLength(x, left, path_length + 1) else
pathLength(x, right, path_length + 1)
}
}
}5、读取数据进行预测
本节定义最终要调用运行的main方法,我把样例数据放在了本地,也可以放到hdfs上,csv格式,已经做好了标准化,概览如下
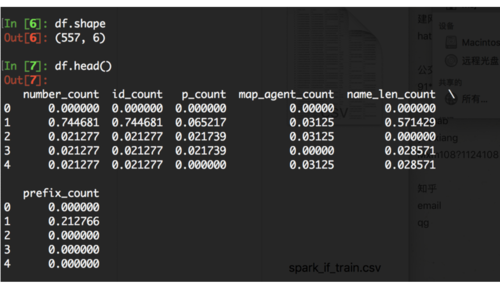
训练数据概览.png
5.1、一些对spark的基本设置
object Runner{
def main(args:Array[String]): Unit ={
Random.setSeed(1337)
val conf = new SparkConf()
.setAppName("IsolationTree")
.setMaster("local")
val sc = new SparkContext(conf) //禁止对输出文件进行压缩
sc.hadoopConfiguration.set("mapred.output.compress", "false")5.2、读入csv数据并预处理,lines为RDD格式,这是spark处理数据的基本单元
val lines = sc.textFile("file:///tmp/spark_data/spark_if_train.csv") //本地路径
val data = //对每一行数据以逗号为分隔符进行拆分,从第二个数据开始取,因为第一个数字是索引
lines
.map(line => line.split(","))
.map(s => s.slice(1,s.length))
val header = data.first() // 取第一行的数据作为列名
// 去掉列名行并将数据转化为double类型
val rows = data.filter(line => line(0) != header(0)).map(s => s.map(_.toDouble))
println("Loaded CSV File...")
println(header.mkString("\n")) // 看一下列名
println(rows.take(5).deep.mkString("\n")) // 看一下前5行数据5.3、进行iforest的构建和对样本的预测
// 构建森林,训练数据rows,森林里树的棵树,这里写10,数据量大的话一般是100
val forest = IsolationForest.buildForest(rows, numTrees=10) // 对每一行数据进行预测
val result_rdd = rows.map(row => row ++ Array(forest.predict(row))) // 将结果存入本地文件
result_rdd.map(lines => lines.mkString(",")).repartition(1).saveAsTextFile("file:///tmp/predict_label") // 看一下前10条数据的预测结果
val local_rows = rows.take(10) for(row <- local_rows){
println("ForestScore", forest.predict(row))
}
println("Finished Isolation")
}
}以上,isolation forest训练部分和预测部分都做好了。
三、部署到spark上并运行
(图片给自己的机器打了码,略丑)
1、基础环境配置
前提1:配置好spark集群,能成功进入下图所示的交互状态。此部分教程自行google~
$ spark-shell
进入spark交互命令行查看是否正常运行.png
前提2:配置好sbt ,用于管理项目依赖,构建项目
参考教程:http://blog.csdn.net/zcf1002797280/article/details/49677881
sbt sbtVersion
查看sbt版本信息确保sbt正确安装.png
2、部署脚本
2.1、将上节代码文件命名为Runner.scala
2.2、创建目录结构
cd ~ mkdir -p mysparkapp/iforest_model/src/main/scala
2.3、将Runner.scala移动到~/mysparkapp/iforest_model/src/main/scala文件夹下
mv Runner.scala ~/mysparkapp/iforest_model/src/main/scala/
2.4、新建配置文件conf.sbt,声明我们项目的名称以及对相关版本的依赖信息
cd ~/mysparkapp/iforest_model vim conf.sbt
在conf.sbt中,添加如下内容,版本信息根据你配置的真实信息来写哦:
name := "IsolationForest"version := "1.0"scalaVersion := "2.11.8"libraryDependencies += "org.apache.spark" %% "spark-core" % "2.2.1"
现在看一下我们的项目结构是否如图所示
find .
查看项目目录结构.png
2.5、将程序打包,仍然在~/mysparkapp/iforest_model下,执行:
sbt package
sbt打包.png
注意黄色箭头指向的文件地址,这是打包好的jar包,供我们稍后提交任务使用。
2.6、正式提交spark任务
在提交spark任务之前,要确保输出目录不存在:
rm -r /tmp/predict_label
然后用spark-submit命令提交任务,需要传入刚刚打包好的jar包路径:
spark-submit --class "Runner" ~/mysparkapp/iforest_model/target/scala-2.11/isolationforest_2.11-1.0.jar
开始运行~~?️
我们打印出了数据的列名、前5条数据、以及前10条数据的异常得分如图所示:
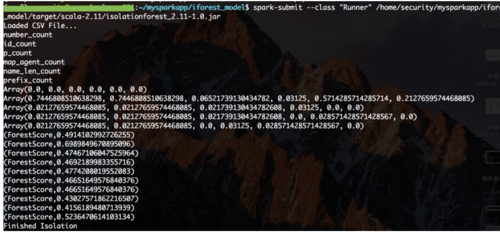
程序执行.png
任务执行完毕,看一下输出文件,图示捞出了前五行,最后一个字段即为预测得分,接下来就可以设定一个阈值,原作论文推荐为0.6,大于阈值的即判定为异常啦。
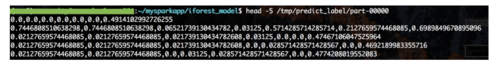
输出文件.png
图中的第二行数据,得分0.69,其他数据得分均为0.5以下,观察一下它前面的字段,比其他数据都要大出很多,确实为一个异常点~
四、小结
isolation forest由多棵树构成,而树的生长过程并不受其他树影响,所以是一个非常完美的适合分布式并行的算法。样例数据和代码都放到了https://github.com/scarlettgin/isolation_spark
作者:双er
链接:https://www.jianshu.com/p/a01a258ee7f2

 随时随地看视频
随时随地看视频




